易基因|RRBS单碱基绘制580种动物的基因组规模DNA甲基化谱:Nature子刊

大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
2023年01月16日,奥地利科学院分子医学研究中心(CeMM)研究团队在《Nat Commun》杂志发表了题为“Comparative analysis of genome-scale, base-resolution DNA methylation profiles across 580 animal species”的研究论文,该研究通过优化版简化基因组重亚硫酸盐测序(RRBS)技术绘制了580种动物(535种脊椎动物,45种无脊椎动物)的DNA甲基化图谱,共生成了2443个基因组规模的多器官DNA甲基化谱。研究构建了脊椎动物和无脊椎动物DNA甲基化组的大量资源,展示了在无参基因组物种中进行无参表观基因组分析的能力,并为脊椎动物进化研究提供了表观遗传学视角。

标题:Comparative analysis of genome-scale, base-resolution DNA methylation profiles across 580 animal species 碱基分辨率下580种动物基因组规模DNA甲基化谱的比较分析
时间:2023.01.16
期刊:Nature Communications
影响因子:IF 17.694
技术平台:RRBS等
样本实验:
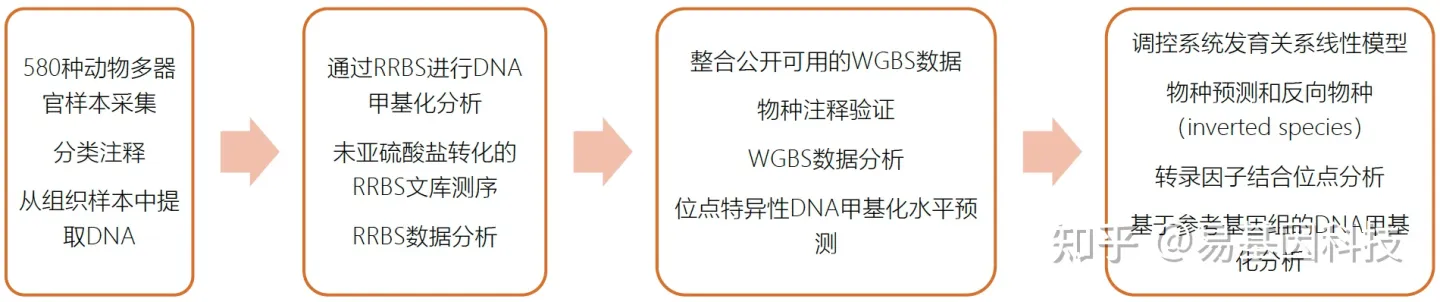
研究摘要:
在脊椎动物进化的广泛背景下,为研究人类基因组之外的DNA甲基化,本研究通过RRBS在535种脊椎动物和45种无脊椎动物中以单碱基分辨率绘制了基因组规模的DNA甲基化谱,涵盖所有脊椎动物类别和几个近端无脊椎动物类别。其中心脏样本和肝脏样本用于物种组织匹配比较,肺、鳃、鳍、脾、脑、淋巴结、肌肉、肾和皮肤等其他组织以物种特异性方式包括在内。样本优先考虑健康成体和平衡雄雌比例,每种物种2-4个。
研究使用优化版简化基因组重亚硫酸盐测序(RRBS)进行DNA甲基化分析。检测区域不仅包括富含CpG的调控区域,也包括外显子、内含子、基因间区域和重复元件等基因组其他区域;检测了基因组CpG位点和非CpG位点的DNA甲基化。为同时研究目前没有公布参考基因组的物种、避免由于可用参考基因组的质量不同而产生的偏差,本研究使用与参考基因组无关的生物信息学方法分析生成RRBS数据集,并在此前三个物种无参和有参分析的头对头比较中验证了这种方法。
本研究完整数据集涵盖580种动物(535种脊椎动物和45种无脊椎动物)的2443个DNA甲基化谱。基于该数据集,研究鉴定出DNA甲基化与脊椎动物和无脊椎动物之间共有潜在基因组DNA序列的定量及预测相关联。研究结果表明了沿着进化轴的两个主要转变:一个在脊椎动物和无脊椎动物之间,另一个在两栖动物和爬行动物之间。同时还研究了DNA甲基化的组织特异性和个体间差异:对于鱼类、鸟类和哺乳动物,组织特异性差异比个体间差异更为显著,但对于无脊椎动物、爬行动物和两栖动物,这两个因素表现出DNA甲基化差异的相似比例。通过分析整个脊椎动物进化过程中心脏和肝脏组织之间差异甲基化区域的转录因子结合位点,结果表明DNA甲基化与组织同一性的高度保守相关联。最后与现有参考基因组的交叉比对鉴定出基因启动子区DNA甲基化的特异性进化趋势。
本研究为脊椎动物进化提供了表观遗传学视角,为揭示DNA甲基化在脊椎动物和无脊椎动物中的作用构建了重要资源。此外研究结果阐明了将表观基因组分析纳入正在进行的所有脊椎动物基因组图谱分析中的可行性和价值,并为揭示DNA序列模式和DNA甲基化的复杂互作如何促进脊椎动物基因组进化提供了新起点。
结果图形
(1)RRBS绘制580种动物的DNA甲基化图谱及脊椎动物进化中全基因组DNA甲基化模式
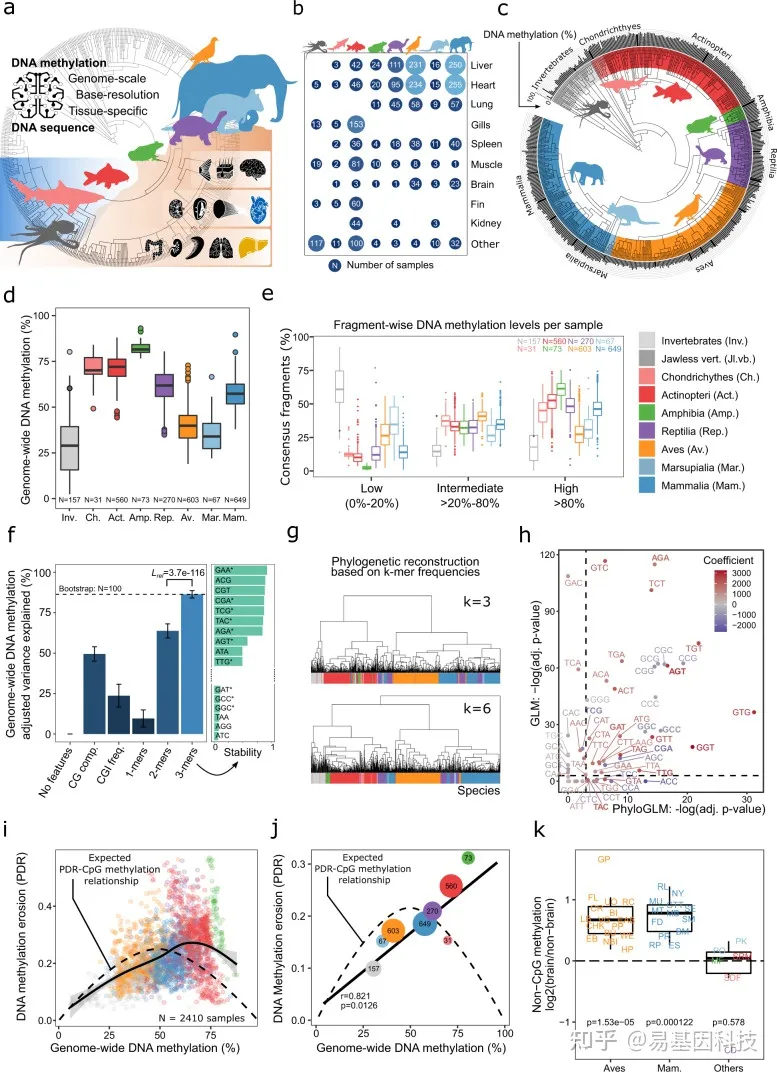
图1:580种动物的DNA甲基化图谱揭示脊椎动物进化过程中基因组和表观基因组之间的整体关联。
跨物种图谱,涵盖580种动物(535种脊椎动物和45种无脊椎动物)的2443个基因组规模的DNA甲基化图谱。动物轮廓表示不同物种:章鱼(无脊椎动物)、鲨鱼(软骨鱼)、鲤鱼(硬骨鱼)、青蛙(两栖动物)、乌龟(爬行动物)、鸽子(鸟类)、袋鼠(有袋动物)、大象(真兽类哺乳动物),器官轮廓表示包括的主要组织(胚层组织)。
每个组织和分类组的分析样本数量气泡图。
每个物种的全基因组DNA甲基化水平条形图(圆圈外的黑条),所有组织和个体的平均值,比对到带注释的分类树上。
所有物种按分类组汇总的全基因组DNA甲基化水平箱线图。
每个物种的共有参考片段百分比箱线图,片段根据其DNA甲基化水平分为三个分类组,包括至少10 reads覆盖片段。
左:由基因组DNA序列特征阐明的物种特异性平均DNA甲基化水平之间的差异百分比条形图。颜色表示平均Akaike信息标准(AIC),根据模型复杂性进行调整。误差线表示基于自举法(100次迭代)平均值的标准偏差。右:使用逐步选择将单个3-mer选择到最终模型的稳定性。*号表示响应的3-mer基于图h中描述的系统发育广义线性模型显示出统计上的显著关联。
基于共有参考片段中3-mer和6-mer频率相似性的物种分层聚类。k-mer长度为4和5的聚类显示出非常相似的结果。
基于具有(x轴)和不具有(y轴)系统发育关系校正的广义线性模型(GLMs)的标准误差散点图,比较3-mer频率和全基因组DNA甲基化水平之间相关性的统计显著性(p值)。
全基因组DNA甲基化水平与DNA甲基化侵蚀(DNA methylation erosion)之间的关系散点图,通过单个样本的“不一致reads比例”(PDR)进行分析。虚线表示数学上预期的关系。实线表示使用R函数geom_smooth拟合到数据的广义加性模型。
全基因组DNA甲基化水平与分类组DNA甲基化侵蚀之间的关系散点图,取相应样本的中位数。虚线表示数学上预期的关系(如图i所示)。实线表示拟合到数据的线性回归模型。两侧显示Pearson相关性及其显著性。
与同一物种中的其他组织相比,大脑中非CpG甲基化水平的对数比率(log-ratios)箱线图。单侧配对Wilcoxon试验评估大脑中非CpG甲基化水平的增加。
(2)脊椎动物和无脊椎动物DNA甲基化的基因组编码
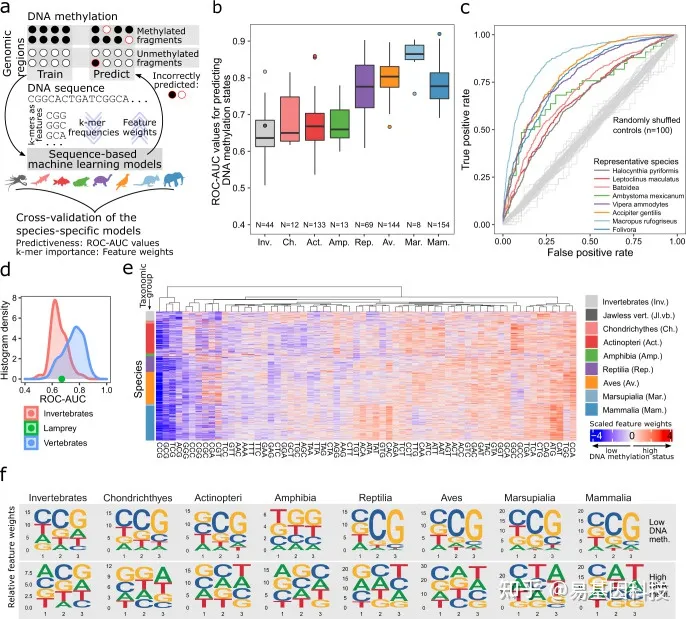
图2:机器学习识别DNA序列和位点特异性DNA甲基化之间的预测关系(“基因组编码”)
基于机器学习方法的示意图,用于从基因组DNA序列预测位点特异性DNA甲基化。
支持向量机(SVM)基于相应基因组DNA序列k-mer频率来预测基因组区域的DNA甲基化水平(高与低)的测试集性能(受试者操作特征曲线下面积,ROC-AUC)箱线图
每个分类组的代表性ROC曲线,所显示物种的ROC-AUC值密切反映对应分类组的平均ROC-AUC值。随机标签数据上训练和评估的ROC曲线接近对角线作为阴性对照(灰色)。
脊椎动物和无脊椎动物的ROC-AUC值的直方图,七鳃鳗(早期无颚脊椎动物)在两种分布之间显示为绿点。
基于SVM的3-mer的特征权重热图,所述SVM training后针对每个物种(由分类树排序)可以预测位点特异性DNA甲基化。
每个分类组跨物种间的3-mer平均特征权重序列标志。序列标志分别显示与低和高DNA甲基化水平相关的3-mer。
(3)DNA甲基化基因组编码的保守和分化
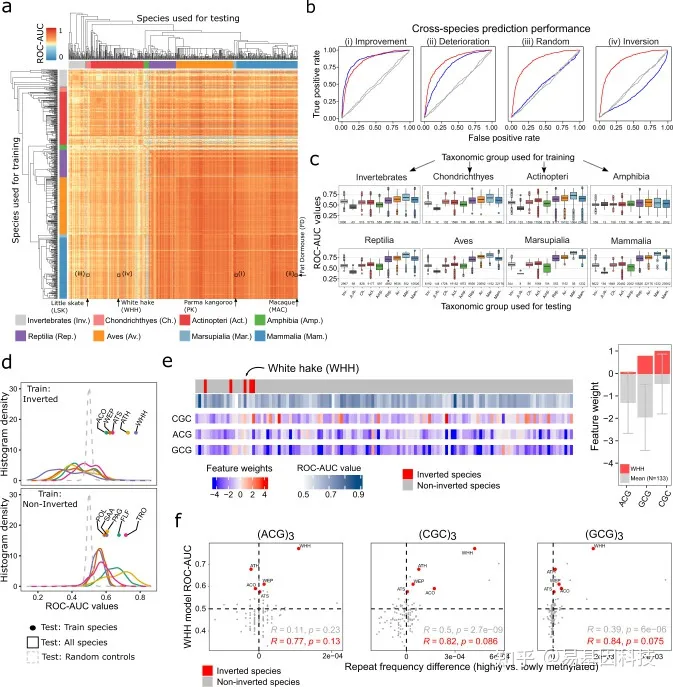
图3:位点特异性DNA甲基化的“基因组密码”在脊椎动物和无脊椎动物中广泛保守。
预测所有物种DNA序列的位点特异性DNA甲基化ROC-AUC值热图。
在肥睡鼠物种(fat dormouse,FD)中训练并在其他物种中测试的分类器的跨物种预测特征结果ROC曲线(从左到右:Parma-wallaby,PK;macaque,MAC;little skate,LSK;white hake,WHH)。在非反向物种中training时,“反向物种(inverted species)”特征比随机预测性能更差。
按training物种(个体图)和test物种(x轴)分类组汇总的物种预测性能(图a的ROC-AUC值)箱线图。
与系统发育相关的非反向物种(下)相比,所有反向鱼类物种(上)的物种预测性能直方图(图a的ROC-AUC值)。反向物种:Atlantic cod, ACO; walleye pollock, WEP; Atlantic salmon, ATS; Atlantic herring, ATH; white hake, WHH. 非反向物种: Pollock, POL; silver arowana, SAA; Pacific grenadier, PAG; onefin flashlightfish, FLF; trout, TRO。
左:反向物种(白鳕鱼,WHH)和所有其他骨鱼(actinopteri)物种(按分类树排序)之间最大差异3-mer的分类器特征权重。右:白鳕鱼相同3-mer的体重与所有其他骨鱼(actinopteri)物种的平均值条形图。误差线表示平均值的标准偏差。
反向物种(白鳕鱼,WHH)中training的分类器跨物种预测性能(y轴)与通过图d加权差分3-mer重复构建的三个9-mer重复(x轴)频率差异之间的关联散点图。>0值表示高甲基化序列中的频率较高,反之亦然。反向物种:大西洋鳕鱼(ACO)、白眼狭鳕(WEP)、白鳕鱼(WHH)、大西洋鲑鱼(ATS)、大西洋鲱鱼(ATH)。虚线表示频率差为0(垂直线),ROC-AUC值为0.5(水平线)。
(4)组织特异性DNA甲基化模式的进化保守

图4:组织特异性DNA甲基化表明DNA甲基化与转录调控和组织特性的高度保守相关。
每个物种的位点特异性DNA甲基化变化百分比散点图,分别由不同分类组的组织(x轴)和个体(y轴)阐明。箭头和p值表示组织和个体解释的方差差异方向和统计显著性,使用双侧成对Wilcoxon检验计算。虚线箭头表示无显著差异,文子云(Word clouds)总结了每个分类组中有助于分析的组织类型频率。
心脏组织和肝脏组织(给定物种内)之间鉴定的差异甲基化区域中转录因子结合位点(TFBS)motif的富集分析示意图。
心脏和肝脏之间差异甲基化片段的TFBS motif富集的聚类热图。每个转录因子(列)颜色表示其是否富含相应物种(行)的心脏(蓝色)或肝脏(黄色)中的低甲基化片段。根据《人类蛋白质图谱》,该热图仅包括每个物种至少有十个显著富集的转录因子和物种,以及心脏或肝脏组织中标准化RNA表达值>1。
图c中鉴定的转录因子的GO注释。
基于图c中鉴定的具有已知结合偏好(甲基化/非甲基化)的转录因子及其具有已知调控作用(激活:绿色;抑制:红色)的直接靶基因构建的基因调控网络。在一种组织类型中偏好低甲基化的转录因子用黄色(心脏)或蓝色(肝脏)表示,而没有表现出这种富集的转录因子以及转录因子靶基因用灰色表示。插图显示心脏和肝脏中FOXO4和EGR1的特异性富集,其对HIF1A的作用相反(FOXO4:激活;EGR1:抑制)。底部图片表示每个分类组中的一个物种,有助于对跨物种的心脏和肝脏的DNA甲基化差异分析。
(5)脊椎动物进化中DNA甲基化的基因中心模式

图5:人类同源基因空间中DNA甲基化的跨物种分析鉴定了启动子甲基化的保守和分化。
基于无参的共有参考片段与注释参考基因组的交叉比对,基因启动子区域DNA甲基化的UMAP表示。样品按分类组表示,匹配的参考基因组为黑色。参考基因组由其UCSC基因组浏览器标识符注释。插图:加扰数据的UMAP表示显示调控分析中缺乏聚类。
基于鸟类和哺乳动物的启动子甲基化数据,使用交叉比对数据集区分心脏和肝脏的随机森林分类器的ROC曲线。实线基于真实数据,虚线基于加扰数据(图a中的插图)。给出真实数据(1)和加扰数据(2)的ROC-AUC值。
心脏与肝脏分类中四种最具预测性基因的基因启动子区域DNA甲基化水平箱线图。
基于心脏和肝脏样本的启动子甲基化数据,使用交叉比对数据集区分鸟类和哺乳动物的随机森林分类器ROC曲线。格式与图b相同。
哺乳动物与鸟类分类中四种最具预测性基因的基因启动子区域DNA甲基化水平箱线图。格式与图c相同
总结:
本研究通过RRBS测序生成的DNA甲基化数据建立了一个规模空前的数据集,并通过对各种动物物种的DNA甲基化保守和分化等不同方面的深入了解,初步阐明了与脊椎动物进化相关的DNA甲基化景观。最值得注意的是,研究发现DNA序列和DNA甲基化在脊椎动物和无脊椎动物物种中表现出广泛的相关性,这些关联在脊椎动物进化过程中逐渐发生变化。研究所生成的数据和分析为研究在人类和动物种群以及各种疾病中的表观遗传异质性提供了进化背景。
关于易基因简化基因组甲基化测序(RRBS)研究解决方案
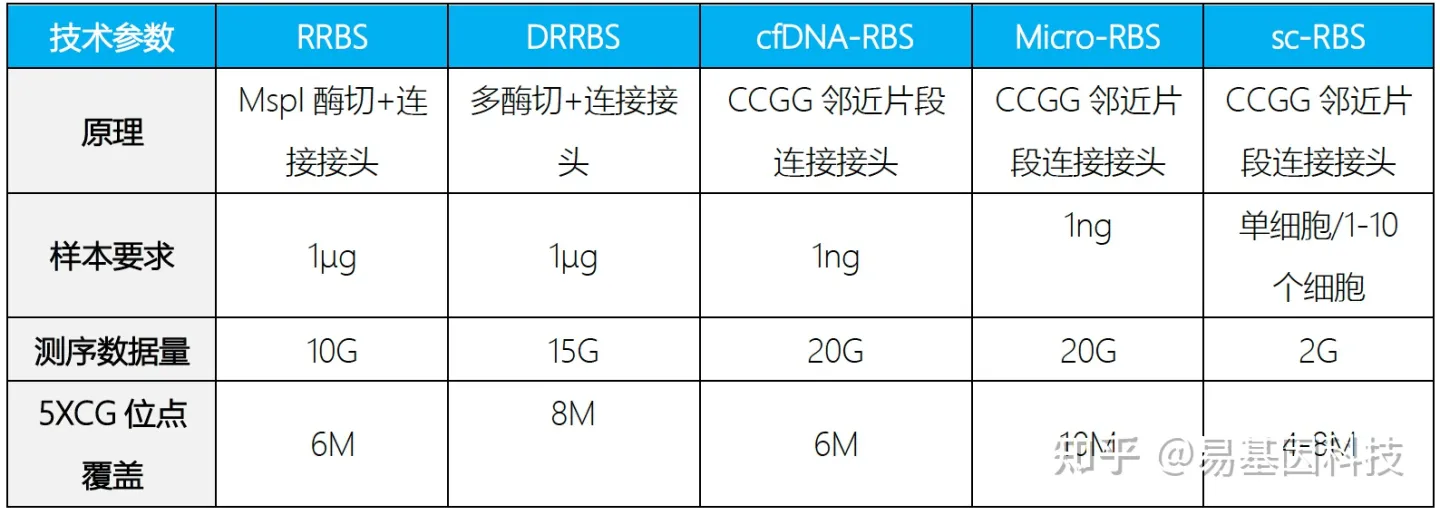
简化甲基化测序(Reduced Representation Bisulfite Sequencing,RRBS)是利用限制性内切酶对基因组进行酶切,富集启动子及CpG岛等重要的表观调控区域并进行重亚硫酸盐测序。该技术显著提高了高CpG区域的测序深度,在CpG岛、启动子区域和增强子元件区域可以获得高精度的分辨率,是一种准确、高效、经济的DNA甲基化研究方法,在大规模临床样本的研究中具有广泛的应用前景。
为适应科研技术的需要,易基因进一步开发了可在更大区域内捕获CpG位点的双酶切RRBS(dRRBS),可研究更广泛区域的甲基化,包括CGI shore等区域。
为助力适用低起始量DNA样本(5ng)量多维度甲基化分析,易基因开发了富集覆盖CpG岛、启动子、增强子、CTCF结合位点的甲基化靶向基因组测序方法:extended-representation bisulfite sequencing(XRBS),实现了高灵敏度和微量样本复用检测,使其具有高度可扩展性,并适用于有限的样本和单个细胞基因组CG位点覆盖高达15M以上。
技术优势:
起始量:100ng gDNA;
单碱基分辨率;
多样本的覆盖区域重复性可达到85%-95%、测序区域针对高CpG调控区域,数据利用率更高;
针对性强,成本较低;
基因组CG位点覆盖高达10-15M,显著优于850K芯片。
应用方向:
RRBS/dRRBS/XRBS广泛应用于动物,要求全基因组扫描(覆盖关键调控位点)的:
队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选
复杂疾病及肿瘤发病机制等甲基化研究
模式动物发育和疾病甲基化研究
易基因科技提供全面的DNA甲基化研究整体解决方案,技术详情了解请致电易基因。
参考文献:
Klughammer J, et al. Comparative analysis of genome-scale, base-resolution DNA methylation profiles across 580 animal species. Nat Commun. 2023 Jan 16;14(1):232.
相关阅读:
技术推介 | 简化基因组甲基化测序(RRBS)研究解决方案
一文读懂|精准简化基因组甲基化测序(RRBS+oxRRBS)分析怎么做
一文看懂|简化基因组DNA甲基化测序(RRBS)实验怎么做
3文一览:简化甲基化测序(RRBS)技术优势及研究成果(医学+物种保护+农学)